| UNITE DE FORMATION | MODULES | OBJECTIFS | INTERVENANT |
|---|---|---|---|
| UF1.1 - Outils numériques et concepts fondamentaux | Extraction et mise en forme des données | familiariser les étudiants avec l'environnement Unix, l'extraction d'information et la manipulation de fichiers de grande taille, l'automatisation de ces traitements |
Christophe Klopp
Anis Djari |
| UF1.2 - Outils numériques et concepts fondamentaux | Stat & R | Présenter les principaux concepts statistiques au travers de R | Elie Maza |
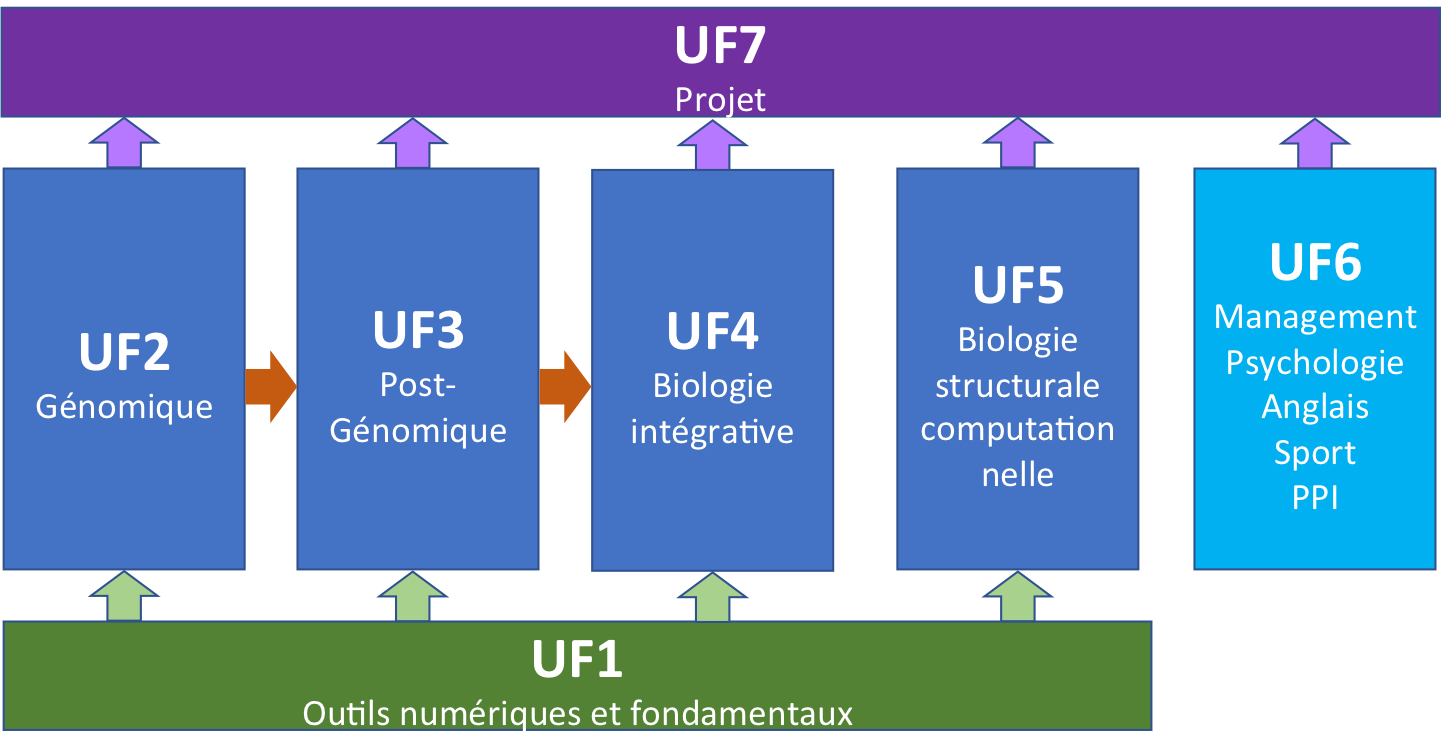
| UF2.1 - Bioinformatique pour la génomique | Assemblage de novo et annotation des génomes | Présenter des techniques d’assemblage des génomes et des transcriptomes et leur annotation structurale et fonctionnelle | Mohamed Zouine |
| UF2.2 - Bioinformatique pour la génomique | Alignement de séquences et recherche de polymorphisme | Apprendre à traiter les séquences issues des NGS pour la recherche de polymorphisme | Mohamed Zouine |
| UF2.3 - Bioinformatique pour la génomique | Bio-informatique pour l’épigénome et le métagénome | Analyser l'épigénomique à différents niveaux | Mohamed Zouine |
| UF3.0 - Post-Génomique | Généralité | Présentation des approches "omiques" |
Brice Enjalbert
Sébastien Déjean |
| UF3.1 - Post-Génomique | Transcriptomique | Présenter les approches visant à quantifier les molécules d’ARN |
Mohamed Zouine
Elie Maza Brice Enjalbert |
| UF3.2 - Post-Génomique | Protéomique | Présenter les approches visant à identifier et à quantifier les protéines à l’échelle génomique |
Brice Enjalbert
David Bouysset |
| UF3.3 - Post-Génomique | Métabolome et Fluxome | Présenter les approches visant à identifier et à quantifier les pools de métabolites |
Brice Enjalbert
Pierre Millard |
| UF4.1 - Biologie intégrative | Intégration statistique des données (avec mixOmics) | Présenter des techniques d’assemblage des génomes et des transcriptomes et leur annotation structurale et fonctionnelle | Ignacio Gonzalez |
| UF4.2 - Biologie intégrative | Modélisation statistique Bayésienne | Présenter une méthodologie permettant de concevoir et de simuler des modèles statistiques complexes. | Christophe Laplanche |
| UF4.3 - Biologie intégrative | Modélisation dynamique d’un système biologique avec régulation | Montrer les principales méthodologies utilisées pour l’analyse des réseaux métaboliques | Cesar Aceves Lara |
| UF5.1 - Biologie structurale computationnelle | Bases théoriques et méthodologiques | Fournir une introduction générale à la modélisation moléculaire |
Isabelle André
Sophie Barbe |
| UF5.2 - Biologie structurale computationnelle | Mise en pratique des méthodes | Mettre en application des méthodes de modélisation moléculaire |
Isabelle André
Sophie Barbe |
| UF7.1 - Projets | Organisation d’une table ronde au forum carrière ENSAT (étudiant ENSAT) | Démarcher une entreprise, d’entretenir un contact professionnel, de travailler efficacement en groupe, de planifier la réalisation d'un évènement. |
Mohamed Zouine
Christophe Laplanche |
| UF7.2 - Projets | Définition du « Défi en bio-informatique » (étudiants ENSAT) | Définir un cahier des charges, de planifier une tâche complexe | Christophe Laplanche |
| UF7.3 - Projets | Réalisation du « Défi en bio-informatique » | Mobiliser des connaissances techniques diverses, de structurer et présenter ses résultats. | Christophe Laplanche |